セルソーター/セルアナライザーの技術情報/導入事例 – 目的のデジタルPCR液滴(エマルジョン)のsortingとターゲットDNAのアンプリコンの精製
【概要】ここ数年、デジタルPCRを用いたDNAやRNAの遺伝子研究の発展は目覚ましい。デジタルPCRの特徴である「絶対定量」を活かし、がん研究や幹細胞研究においてレアミューテーションの検出やCNV (Copy Number Variation)、 遺伝子発現、メチル化解析など年々数々の研究成果が報告がされ、早期診断ツールとしての有用性も示唆されている。その一方、結果の解釈がユーザー依存型であり、設定条件によって結果が大きく左右されるなどの問題点も挙げられる。結果の解釈がユーザー依存的であることの回避を考えると、どこまでが陽性なのかという結果を明確にすることが重要になってくる。そこを追及するためにはデジタルPCR同等エマルジョンで蛍光を示すサンプルを回収し、実際に目的のものなのか確認する必要がある。
【目的】目的の変異配列のみを含むデジタルPCRエマルジョンのソーティングができないか検討した。
【方法】デジタルPCRの液滴サンプルは、 Bio-Rad QX200™システムの方法に従ってサンプル作成を実施した。DNAは、野生型配列と変異型配列を9:1の割合で混合し準備した。プライマー、変異型特異的なTaqMan®プローブ(FAM)とDNAサンプルを2xSuper Mix for probes(BioRad社)に規定量を加えて、液滴形成およびPCR反応を行った。On-chip Sort を用いてシースにはオイルを使用し、PCR反応後の液滴(40μℓ)のソーティングを実施した。
リアルタイムPCRによる確認はソーティングした液滴からのDNAの精製は、油層をクロロフォルムで溶解しDNA精製用スピンカラムを用いて行った。精製したDNAは、リアルタイムPCR(rtPCR)法を用いて目的断片の有無を確認した。(リアルタイムPCRの条件はデジタルPCR時と同じ条件で行った。)
【結果・考察】TaqMan®FAMは、FL2で検出し、変異型はTaqMan®FAMポジティブとして検出された。On-chip SortでDNAの野生型と変異型と9:1で混合した結果が正確に検出された (Fig.1, Fig.2)。
目的変異を含む液滴のみを取得し、得られた液滴から増幅DNA(アンプリコン)を精製することが出来た。またリアルタイムPCRによる確認では目的断片の存在が確かめられた(Fig.3)。
これにより、デジタルPCR同等エマルジョンを回収するのに有用なソーターであることが示唆された。また On-chip Sortは遺伝子配列の決定など次世代DNAシーケンサーが必要とする高純度サンプルの作製としても有用であると考えられる。
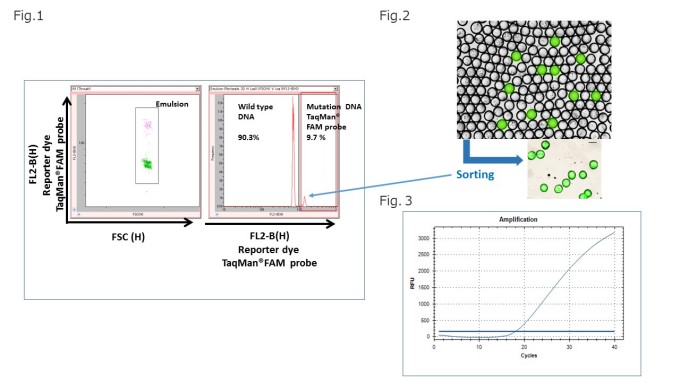
Fig.1 Bio-Rad QX200™システム( On-chip Droplet Generatorでも可能)を用いて約100μmエマルジョンを作製しTaqManプローブにおけるPCR反応後にOn-chip Sortを用いて検出・分取した。
Fig.2 On-chip Sortを用いたソーティング前とソーティング後のエマルジョン写真。約10%の変異型陽性エマルジョンをほぼ100%回収した。
Fig.3 回収したエマルジョン(Fig.2)を破壊・精製し、再度RT-PCRにおいて目的産物の確認をした。