セルソーター/セルアナライザーの技術情報/導入事例 – 薬剤セレクションが不要となるノックアウト細胞株樹立 (CRISPR/Cas SystemとOn-chip SPiSを組み合わせたゲノム編集)
【概要】ゲノム編集ツールの一つとしてCRISPR/Cas Systemがある。このシステムはPAM配列、gRNAとCas9タンパク質が形成する複合体を利用しDNA二本鎖の削除、置換、挿入などが可能になるツールである。このシステムを使用して目的のゲノム編集をした細胞をクローン化するとき純化回収が必要になるが、今回On-chip SPiSを使用して純化培養をおこなった。
(本研究は国内当社機器導入施設のお客様の協力で実施した)
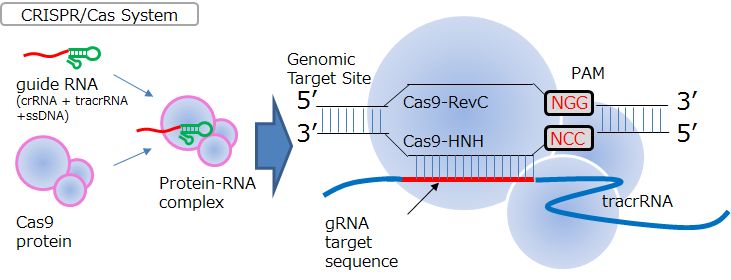
CRISPR/Cas Systemの概要
CRISPR/Cas Systemは、バクテリアの免疫機構として知られています。ウイルスなどの外来DNAがバクテリアのゲノム中にあるCRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)と呼ばれる座位に挿入されることで、免疫記憶として機能しています。詳細には、CRISPR-associated(Cas)9はcrRNA(CRISPR RNA)とtracrRNA(trans-activating crRNA)のヘアピン構造を模倣したガイドRNA(gRNA)に導かれて配列特異的に結合し Casヌクレアーゼにより二本鎖DNAを切断します。この特異性はgRNAによって規定されていてgRNAはPAMモチーフと呼ばれるNGG配列が必要となります。CRISPR/Cas9システムによる配列特異的DNA切断はCas9ヌクレアーゼとgRNAによるもので非常にシンプルになっています。またバクテリア免疫機構が起源ではあるが、多くの生物種で高効率の切断活性を示しますことが知られています。更に作成する上での制限はgRNAにおいてNGG配列だけであることから、デザイン上の自由度が非常に高くoff-targetの危険性の少ない領域に設計することが容易である特徴もあり、ゲノム編集分野で注目されている手法の一つです。
【準備】
細胞導入及び細胞培養
Lipofectamine® RNAiMAX Transfection Reagent (ThermoFisher, Cat.13778030)
Alt-R CRISPR crRNA(IDT), Alt-R CRISPR tracrRNA(IDT)
プレートコーティング用コラーゲン(新田ゼラチン, Cellmatrix)
gDNA抽出及びノックアウト検出
NucleoSpin® 8 Tissue(TaKaRa, Cat.740740)
ddPCR用検出probe(FAM:target用、HEX:リファレンス用BCHDKA)(IDT)
QX200 Droplet Digital PCR(BioRad), ddPCR supermix for Probes(No dUTP)
(BioRad, Cat.186-3023)
細胞分注
On-chip SPiS(On-chip Biotechnologies),
SPiS用1000μlチップ ブルー(WATSON, Cat.110-502B),
SPiS用384分注ピペット(Biotec, Cat. BST5-384S)
細胞
Cas9安定発現HEK293細胞株(協力頂いたお客様のラボで樹立)
【操作】
①Cas9 安定発現HEK293細胞株にLipofectamine RNAi maxを使用してcrRNA+tracrRNA complexをトランスフェクションした。
②72hrs後に細胞を回収して,40umのフィルターを通した。
③On-chip SPiSを使用して細胞を48well分交互に4枚(計192well)コラーゲンコートした96well plateに撒いた。
(分注→スピンダウン後CO2インキュベーターに出来るだけ早く入れて細胞生着しやすいようにした)
④2週後増えた細胞を回収し,gDNAをNucleospin 8 tissue kitを使用して精製した。
⑤gDNAをテンプレートとしてdroplet digital PCRで目的配列のノックアウトの有無を確認した。
(Target probeにFAM, Reference probeにHEXをそれぞれ使用し、ddPCR supermix for Probes(No dUTP)を用いてPCRを実施し,QX200 Droplet Digital PCRにて解析)

【結果】
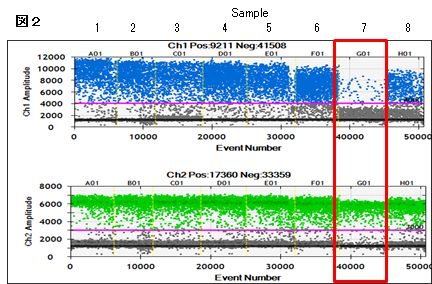
トランスフェクション後、On-chip SPiSにて192ウェル撒いた(図1A)。そのうち生着が認められたもの(図1B)が67ウェル、2週間後細胞が十分に増殖していたもの(図1C)が28ウェルであった。2週間後細胞が十分に増殖したウェルのみddPCR (droplet digital PCR)で目的物のノックアウトを確認した(図2)。28ウェル中3ウェルでノックアウトを確認した。
本確認方法では目的配列プローブを使用してddPCRをかけて増加してこないものをノックアウトしたと判断している。
下の緑のドットがReference(BCHDKA)、上の青のドットがターゲットのプローブを使用したときの結果である。

【まとめ】
本結果からトランスフェクション後に薬剤セレクション不要でノックアウト細胞株を樹立できることがわかった。
しかし生着および増殖してくるウェル数が減少することから、シングルセル培養のような少数細胞培養からの細胞増殖は培養条件の最適化が課題としてあることもわかった。この技術をベースにしてノックアウトのみならずノックイン細胞株の作製や薬剤セレクションをしたくない再生医療分野におけるゲノム編集等に役立たせたい。
今回は編集効率の点からCas9を安定発現する細胞を用いたが、今後GFP-Cas9を一過性に発現する細胞をOn-chip Sortで分離し同様の検討を行うことで、より野生型に近い細胞獲得を目指したいと考えている(図3)。

細胞ダメージが少ないOn-chip SortとOn-chip SPiSの組み合わせで
シングルセル培養が難しいiPSや神経細胞などのゲノム編集細胞取得が可能となる!?
(CRISPR/Cas System参考文献)
・Deltcheva et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature 471(7340):602-7 (2011).
・M. M. Jinek, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012).
・L. A. Marraffini, E. J. Sontheimer, Self versus non-self discrimination during CRISPR RNA-directed immunity. Nature 463, 568 (2010)
・Wang et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell 153(4):910-8. (2013)
・Cong, L., et al., Multiplex genome engineering using CRISPR/Cas systems. Science 2013; 339(6121):819-23.
・Mali, P., et al., RNA-guided human genome engineering via Cas9. Science 2013; 339(6121):823-6.